Infektions- und tumorbiologische Prozesse
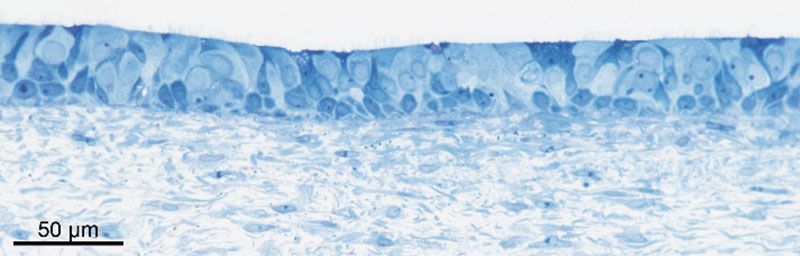
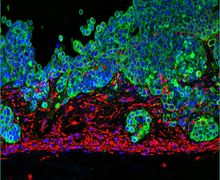
3D-Gewebemodelle, die an unserem Lehrstuhl entwickelt wurden, werden für die Forschung in dieser Arbeitsgruppe herangezogen. Die Gruppe verwendet und entwickelt diese Gewebemodelle weiter unter dem besonderen Aspekt eines breiten Spektrums an Fragestellungen zu Pathomechanismen von Infektions- und Krebserkrankungen.
Graduiertenkolleg 3D-Infekt (GRK 2157)
Unsere Forschungsaktivitäten sind zentraler Bestandteil im Rahmen des Graduiertenkolleg 3D-Infekt (GRK 2157), welches die Deutsche Forschungsgemeinschaft fördert. Hier wird der Einsatz von humanen 3D-Gewebemodellen für die Untersuchung mikrobieller Infektionen durch verschiedene Humanpathogene untersucht, wie Salmonellen-Infektionen auf Darmmodellen oder Trypanosomeninfektionen auf 3D-Hautmodellen.
Erforschung von Atemwegsinfektionen
Im Mittelpunkt eines der Unterprojekte im GRK 2157 steht beispielsweise die Erforschung der Interaktionen von Bordetella pertussis, dem Erreger des Keuchhustens, mit dessen Wirtsgewebe, der menschlichen Atemweg-Schleimhaut. In enger Zusammenarbeit mit dem Lehrstuhl für Mikrobiologie der Universität Würzburg (Professor Dr. Roy Gross) und der Tschechischen Akademie der Wissenschaften (Prof. Peter Sebo) untersucht die Arbeitsgruppe die Auswirkungen des Adenylatzyklasetoxins – abgekürzt ACT – auf Ephitelzellen der menschlichen Atemwege unter 2D- und 3D-Kulturbedingungen.
Aktuell werden die Modelle auch genutzt, um gemeinsam mit der Arbeitsgruppe von Professor Dr. Jochen Bodem am Institut für Virologie der Universität Würzburg definierte Substanzen auf ihre Wirksamkeit gegen SARS-CoV-2, das Virus, das für die Corona-Pandemie des Jahres 2020 verantwortlich ist, zu untersuchen.
Onkologische Therapieentwicklung
In ihrer Forschungsarbeit geht die Arbeitsgruppe auch der Wirksamkeit onkologischer Medikamente nach. Dazu zählt neben der allgemeinen Wirkstofftestung der Einsatz von zielgerichteten Medikamenten, die bei bestimmten Biomarker-Mutationen helfen, oder einer Kombination mit anderen Wirkstoffen, sowie auch von immuntherapeutischen Ansätzen wie CAR-T-Zellen in soliden Tumorgewebemodellen (Kollaboration mit Dr. Michael Hudecek, Medizinische Klinik II, UKW). Diese Modelle können auch durch Zellen der Tumorumgebung erweitert werden. Bisher wurden Modelle für Lungen-, Darm- und Brustkrebs etabliert, weitere befinden sich in der Entwicklung.
In Kooperation mit dem Lehrstuhl für Bioinformatik (Professor Dr. Thomas Dandekar) werden bioinformatorische Netzwerke und in silico-Analysen rechnergestützt für die Biomarker-geleitete Präzisionsmedizin durchgeführt, zum Beispiel des tumorrelevanten EGFR-Signalwegs. Dies ermöglicht es, Wirkmechanismen systemisch besser zu verstehen und so maßgeschneiderte Therapien für die einzelne Patientin und den einzelnen Patienten zu finden.
Einsatz als Alternativen zu Tierversuchen
Die Tumormodelle wurden gemeinsam mit der systembiologischen in silico-Analyse über drei Jahre in dem BMBF-Verbundprojekt Remis 3R als Alternative zu Tierversuchen eingesetzt und gefördert. Des Weiteren führten diese Gewebemodelle schon konkret zu dem Ersatz von Tierversuchen wie in hochrangigen Publikationen im Bereich der allgemeinen Wirkstofftestung und bei der CAR-T-Zellen-Testung gezeigt.