Superhochauflösung in der Fluoreszenzmikroskopie ermöglicht winzige Strukturen biologischer Proben in unglaublicher Detailgenauigkeit zu betrachten. Trotz ihres Potenzials bleibt die Erfassung hochmoderner, superhochaufgelöster Bilder aufgrund des erforderlichen technischen Fachwissens, der zeitintensiven Verfahren und der komplexen Analyse eine Herausforderung.
In dieser Studie präsentiert die Neurologie gemeinsam mit dem Rudolf-Virchow-Zentrum – Center für Integrative und Translationale Bildgebung (RVZ) ein skalierbares Open-Source-Software-Toolkit, das die Bilderfassung mit dSTORM automatisiert. Durch die Nutzung von Deep Learning zur Segmentierung kann das Toolkit Objekte in verschiedenen biomedizinischen Proben präzise identifizieren und gezielt anvisieren, selbst solche mit geringem Kontrast. Diese Automatisierung beschleunigt die Workflows dieser Bildgebung erheblich. Durch die Bereitstellung einer breit zugänglichen, benutzerfreundlichen Lösung können Forscherinnen und Forscher verschiedener Disziplinen die Leistungsfähigkeit der Super-Resolution-Mikroskopie nutzen, ohne eine umfangreiche Spezialausbildung zu benötigen.
Das eigenständige Programm, das in der Fachzeitschrift Biophysical Reports präsentiert wird und sogar den Titel erhalten hat, ermöglicht die zuverlässige Segmentierung biomedizinischer Bilder und übertrifft bestehende Lösungen. Integriert in die Bildgebungs-Pipeline verarbeitet es hochaufgelöste Daten in Minutenschnelle und reduziert so den manuellen Arbeitsaufwand. Anhand biologischer Beispiele wie Mikrotubuli in Zellkulturen und dem βII-Spektrin in Nervenfasern zeigen Janis T Linke und Katrin Heinze vom RVZ gemeinsam mit Luise Appeltshauser und Kathrin Doppler aus der Neurologie, dass der Ansatz die superauflösende Bildgebung schneller, robuster und benutzerfreundlicher macht, auch für Mikroskopie-Laien. Dies erweitert die Anwendungsmöglichkeiten in der Biomedizin, einschließlich Hochdurchsatz-Experimenten.
Janis T Linke, Luise Appeltshauser, Kathrin Doppler, Katrin G Heinze. Deep learning-driven automated high-content dSTORM imaging with a scalable open-source toolkit. Biophys Rep (N Y). 2025 Jun 11;5(2):100201. https://doi.org/10.1016/j.bpr.2025.100201. Epub 2025 Feb 28. PMID: 40023500; PMCID: PMC11986538.
Zur Publikation bei PubMed
Titelbild: Künstlerische Komposition autonom aufgenommener dSTORM-Bilder von farbstoffbeschichteten Mikrokugeln (SpheroRulers), welche die im Vergleich zu einem herkömmlichen Weitfeldbild erreichte verbesserte räumliche Auflösung veranschaulichen.
Erfolgreiche Kooperation von Janis Linke und Katrin G Heinze vom RVZ und Luise Appeltshauser und Kathrin Doppler aus der Neurologischen Klinik (v.l.n.r.)
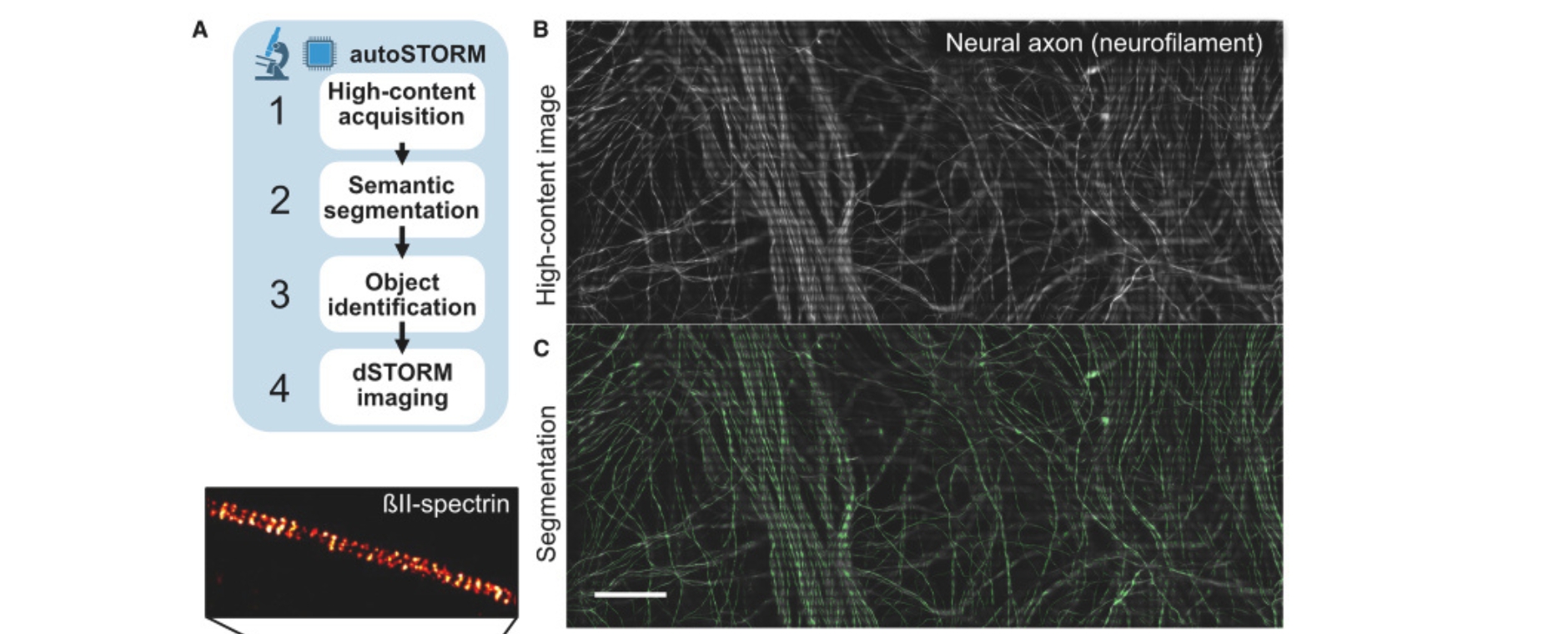
Automatisierte STORM-Erfassung für High-Content-dSTORM-Daten: Durch die Integration von Deep Learning in die Superauflösungsbildgebung können Bilder völlig autonom erfasst werden. (A) Die Erfassung erfolgte nach einem automatischen vierstufigen Protokoll: High-Content-Bilderfassung, gefolgt von semantischer Bildsegmentierung, gefolgt von Objektidentifizierung und schließlich dSTORM-Bildgebung. (B und C) Zur Bildgebung von βII-Spektrin in Nervenaxonen wurden die Axone in einem separaten Farbkanal identifiziert, der für Neurofilamente gefärbt war. (B) Das High-Content-Bild zeigt viele Nervenaxone in einer DRG-Neuronenkultur. Maßstab 100 μm. (C) Das DNN konnte diese Axone für die spätere automatisierte Bildgebung segmentieren (grüne Überlagerung).
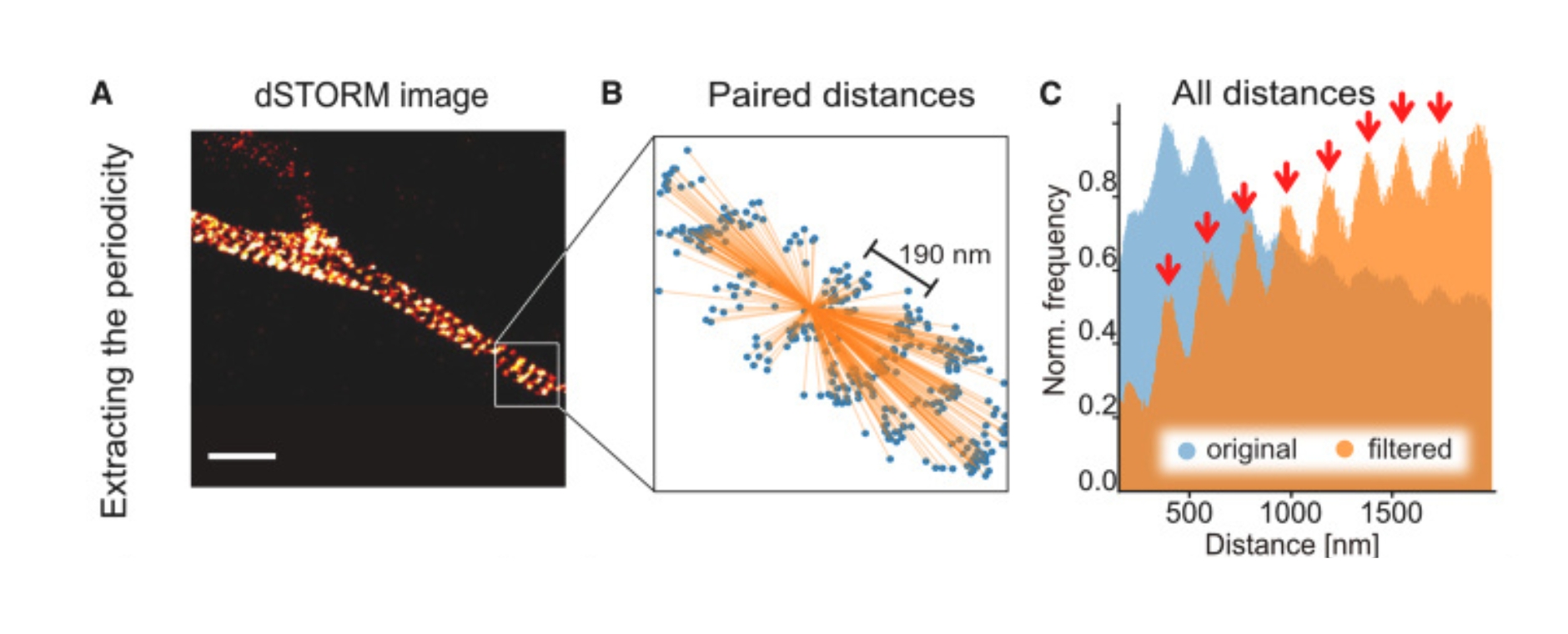
Schnelle Ermittlung axonaler Periodizitäten: Zur schnelleren Ermittlung axonaler Periodizitäten hat das Team eine Erweiterung entwickelt, die nicht von ROI-Auswahlen oder korrekt ausgerichteten Bildausschnitten abhängig ist. (A) dSTORM-Bild von βII-Spektrin in neuronalen Axonen (B) Streudiagramm aller erkannten Emitter in einem zugeschnittenen Bereich von (A) (blau), überlagert mit allen paarweisen Abständen eines Emitters (orange). (C) Histogramm aller paarweisen Abstände aller Emitter von Bild (A) (blau). Gefiltert durch eine Spline-Anpassung an den Verlauf des Axons werden die Peaks deutlich, die die periodische Anordnung der Emitter darstellen (orange). Die Peaks sind mit roten Pfeilen markiert. 190,6 nm.