Eine Polyneuropathie ist eine Erkrankung, bei der mehrere periphere Nerven gleichzeitig geschädigt sind, was zum Beispiel Kribbeln, Taubheitsgefühle, Schmerzen oder Muskelschwäche meist an Händen und Füßen verursacht.
Patientinnen und Patienten, die Anti-Contactin-1-Autoantikörper bilden, leiden häufig zusätzlich an einer Glomerulonephritis, eine entzündliche Erkrankung der Nieren, bei der die feinen Filtereinheiten (Glomeruli) geschädigt werden, sodass unter anderem Eiweiß oder Blut in den Urin gelangen und die Nierenfunktion beeinträchtigt sein kann. Auch Diabetes mellitus tritt bei ihnen gehäuft auf. Die unterschiedlichen klinischen Verläufe und Begleiterkrankungen könnten auf verschiedene Epitope der zugrunde liegenden Autoantikörper zurückzuführen sein. Epitope sind Molekülabschnitte, an denen das Immunsystem gezielt bindet.
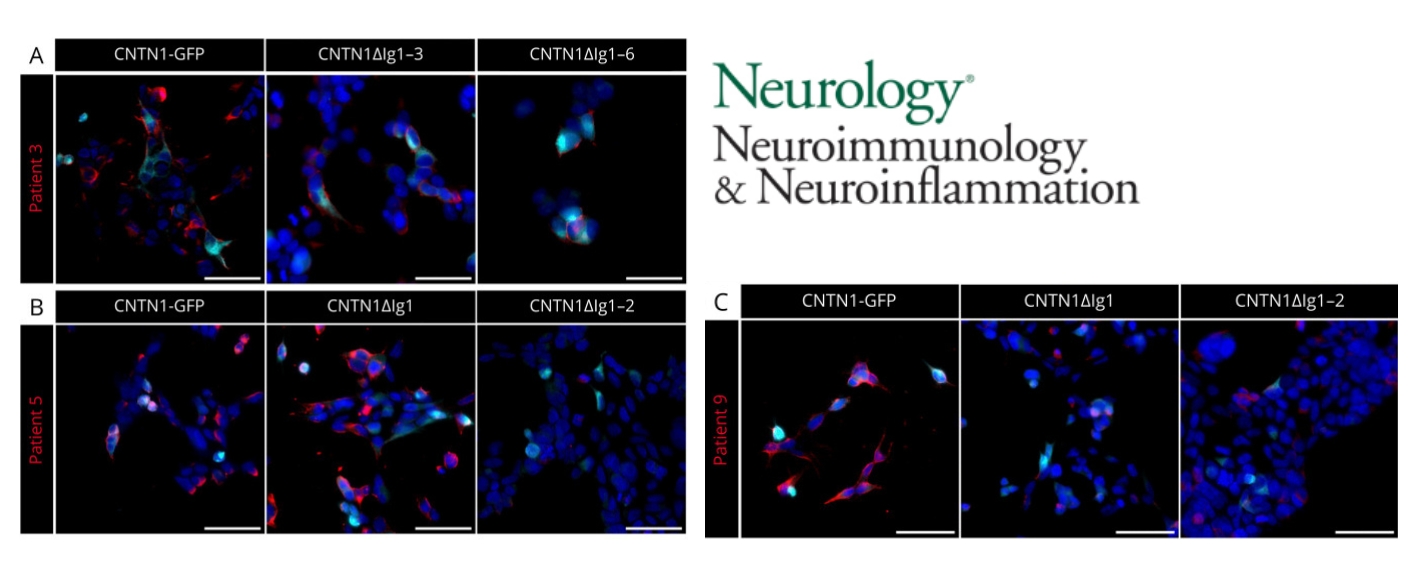
In dieser Studie untersuchten die Arbeitsgruppen von Prof. Dr. Carmen Villmann (Institut für klinische Neurobiologie), Prof. Dr. Kathrin Doppler (Neurologische Klinik) und Dr. Hans Maric (Rudolf-Virchow-Zentrum) über Bindung an Deletionsmutanten und Peptid-Microarrays, ob verschiedene Epitope mit bestimmten klinischen Phänotypen assoziiert sind. Die Forschenden fanden heraus, dass dies tatsächlich der Fall ist. Die Studie zeigt, dass die Erkrankung heterogener ist als bislang angenommen. Das Vorliegen verschiedener Epitope innerhalb der Erkrankung deutet auf verschiedene Auslöser der Autoimmunreaktion und verschiedene Effekte der Autoantikörper im Gewebe hin. Die Bestimmung der Epitope könnte somit ein Prognosefaktor für die Erkrankung des einzelnen Patienten sein.
Publikation
Julia Grüner, Ivan Talucci, Carolin Kurth, Markus Bayer, Luise Appeltshauser, Andreas Steinbrecher, Liis Väli, Sabine Ulrike Vay, Alexander Grimm, Mario Fuchs, Albrecht Günther, Christian Geis, Claudia Sommer, Hans Michael Maric, Carmen Villmann, Kathrin Doppler. Distinct Epitopes Are Associated With Clinical Phenotypes in Autoimmune Nodopathies With Anti-Contactin1 Autoantibodies. Neurol Neuroimmunol Neuroinflamm. 2026 Jan;13(1):e200507. doi: 10.1212/NXI.0000000000200507. Epub 2025 Nov 17. PMID: 41248448; PMCID: PMC12624422.
Zur Publikation



